Simulation numérique de l'étalement cellulaire P. Cañadas, B. Maurin, H. Baudriller
Soumis le 31 Jan 2009 par patrick cañadas
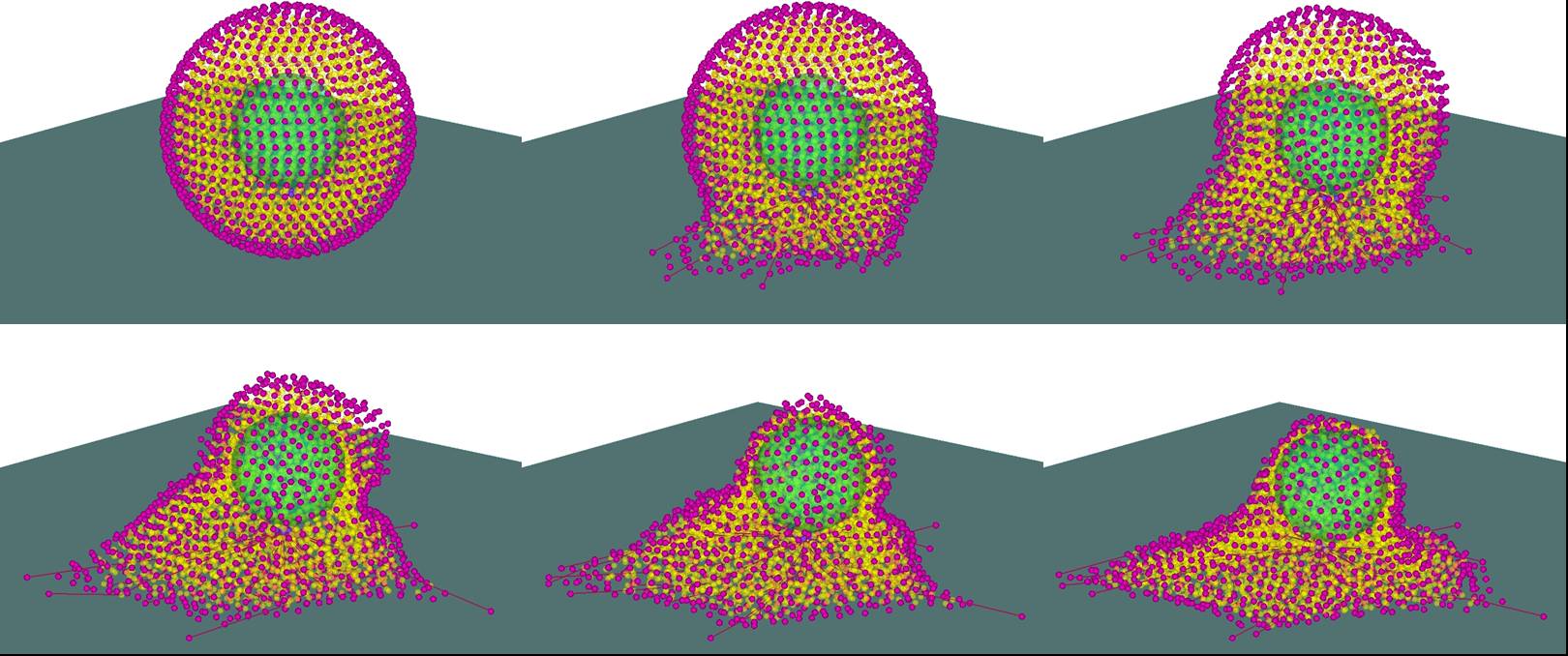
Cette image représente les étapes d'un film (http://www.lmgc.univ-montp2.fr/~canadas/movie/) issu de la simulation numérique 3D de l'adhésion et de l'étalement d'une cellule eucaryote animale sur un substrat plat.
Les outils de base sont issus de la mécanique des milieux divisés ; ici un milieu granulaire particulier : en vert un gros grain central représentant le noyau, en jaune des petits grains représentant des cross-linker, les grains en rose représentant des protéines (trans-)membranaires dont certaines, aléatoirement disposées, représentent des complexes d'adhésion de type intégrines. Le susbtrat est simulé par un plan (ici tronqué, en gris-vert). Tous ces grains sont interconnectés entre eux via des interactions mécaniques de type "contact", mais ici "à distance" (un peu comme des ressort virtuels). A l'issue de cette simulation, il est possible de visualiser les réseaux d'interactions de type "tension" et de type "compression" (cf. images suivantes)
